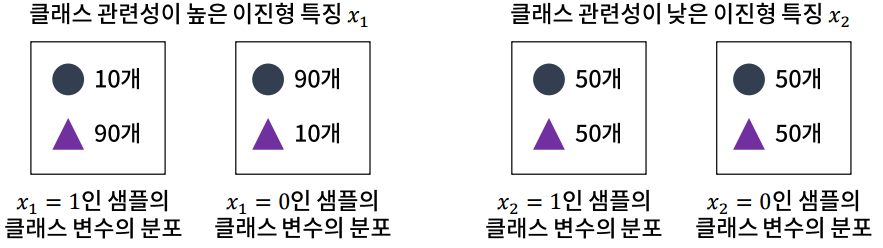
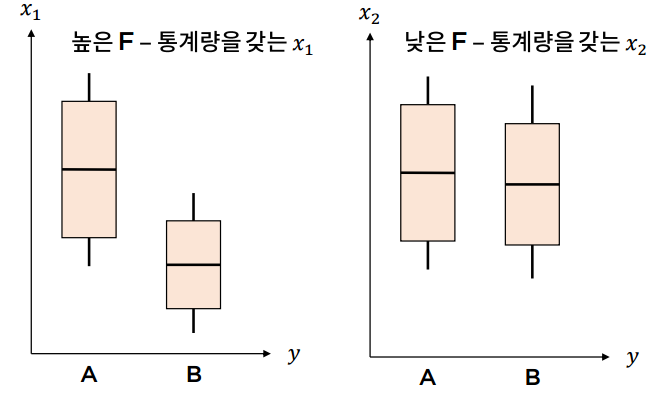
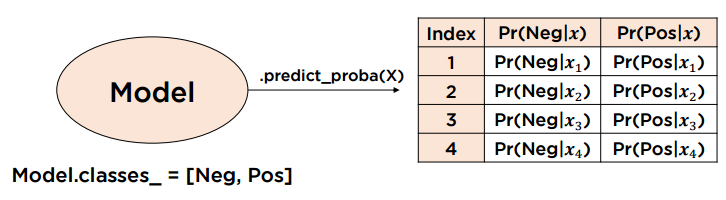
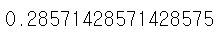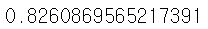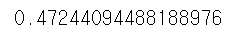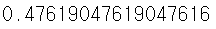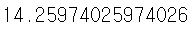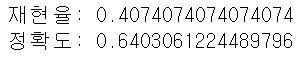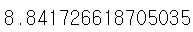지도학습이란?
지도학습은 명확한 정답이 주어진 데이터를 먼저 학습한 뒤 미지의 정답을 예측하는 방식
이때 학습을 위해 주어진 데이터 세트를 학습 데이터 세트, 머신러닝 모델의 예측 성능을 평가하기 위해 별도로 주어진 데이터 세트를 테스트 데이터 세트로 지칭함
하이퍼 파라미터란?
하이퍼 파라미터는 머신러닝 알고리즘별로 최적의 학습을 위해 직접 입력하는 파라미터들을 통칭하며, 하이퍼 파라미터를 통해 머신러닝 알고리즘의 성능을 튜닝할 수 있음
붓꽃 품종 예측하기
추후에 배우게 될 의사결정트리(Decision Tree)를 활용해 붓꽃의 품종을 분류하는 모델을 만들어 머신러닝의 흐름을 파악해보자!
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score# 붓꽃 데이터 세트 로딩
iris = load_iris()
# 피처와 레이블
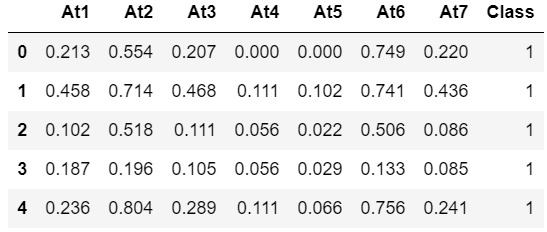
iris_data = iris.data
iris_label = iris.target학습 데이터로 학습된 모델이 얼마나 뛰어난 성능을 가지는지 평가하기 위해 학습/테스트 데이터 분리함
# 학습/테스트 데이터 분리
x_train, x_test, y_train, y_test = train_test_split(iris_data, iris_label,
test_size=0.2, random_state=11)# DecisionTreeClassifier 객체 생성
dt_clf = DecisionTreeClassifier(random_state=11)
# 학습
dt_clf.fit(x_train, y_train)
# 예측
pred = dt_clf.predict(x_test)
# 평가
print('예측 정확도:{0:.4f}'.format(accuracy_score(y_test, pred)))예측 정확도:0.9333사이킷런 프레임워크 익히기(지도학습:분류, 회귀)
●사이킷런은 ML 모델 학습을 위해 fit(), 학습된 모델의 예측을 위해 predict() 메서드 제공
●분류 알고리즘은 Classifier, 회귀 알고리즘은 Regressor로 지칭 → 두 개를 합쳐서 Estimator라고 부름
●cross_val_score와 같은 평가함수, GridSearchCV와 같은 하이퍼 파라미터 튜닝을 지원하는 클래스는 Estimator를 인자로 받음

사이킷런의 주요 모듈

Model Selection 모듈 소개
1. 학습/테스트 데이터 세트 분리 - train_test_split()
sklearn.model_selection의 train_test_split()함수

●첫 번째 파라미터로 피처 데이터 세트 입력
●두 번째 파라미터로 레이블 데이터 세트 입력
●test_size: 전체 데이터에서 테스트 데이터 세트 크기를 얼마로 샘플링할 것인가를 결정함. 디폴트는 0.25, 즉 25%임
●train_size: 전체 데이터에서 학습용 데이터 세트 크기를 얼마로 샘플링할 것인가를 결정함. test_size parameter를 통상적으로 사용하기 때문에 train_size는 잘 사용되지 않음
●shuffle: 데이터를 분리하기 전에 데이터를 미리 섞을지를 결정함, 디폴트는 True임
●random_state: 호출할 때마다 동일한 학습/테스트용 데이터 세트를 생성하기 위해 주어지는 난수값임, train_test_split()은 호출 시 무작위로 데이터를 분리하므로 random_state를 지정하지 않으면 수행할 때마다 다른 학습/테스트 용 데이터를 생성함
하지만 이 방법 역시 과적합(Overfitting)이라는 약점이 존재함
여기서 과적합이란 모델이 학습 데이터에만 과도하게 최적화되어, 실제 예측을 다른 데이터로 수행할 경우에는 예측 성능이 과도하게 떨어지는 경우를 말함, 즉 고정된 학습 데이터로 학습하고 고정된 테스트 데이터를 통해 모델의 성능을 확인하고 수정하는 과정을 반복하면, 결국 내가 만든 모델은 테스트 데이터에만 잘 동작하는 모델이 된다. 이 경우에는 테스트 데이터에 과적합되어 다른 실제 데이터를 가지고 예측을 수행하면 엉망인 결과가 나와버림
→ 이를 해결하기 위해 교차검증을 사용
2. 교차검증
간략하게 설명하자면 본고사를 치르기 전에 모의고사를 여러 번 보는 것임. 즉, 본고사가 테스트 데이터 세트에 대해 평가하는 거라면 모의고사는 교차검증에서 많은 학습과 검증 세트에서 알고리즘 학습과 평가를 수행하는 것

(원칙적으로 train_test_split으로 학습과 테스트 데이터 세트로 분리 후 학습 데이터를 학습과 검증 데이터 세트로 나눠 1차 평가)
→교차검증은 별도의 여러 세트로 구성된 학습 데이터 세트와 검증 데이터 세트에서 학습과 평가를 수행하는 것
⇛ML모델의 성능 평가는 교차 검증 기반으로 1차 평가를 한 뒤 최종적으로 테스트 데이터 세트에 적용해 평가하는 프로세스이다.
(iris데이터는 데이터 세트가 적기 때문에 교차검증이 어떻게 적용되는지를 살펴보기 위해서 학습 데이터에서 검증 데이터 세트를 안나누고 전체 데이터 세트로 교차검증을 수행했다. GridSearchCV는 원칙대로 적용)
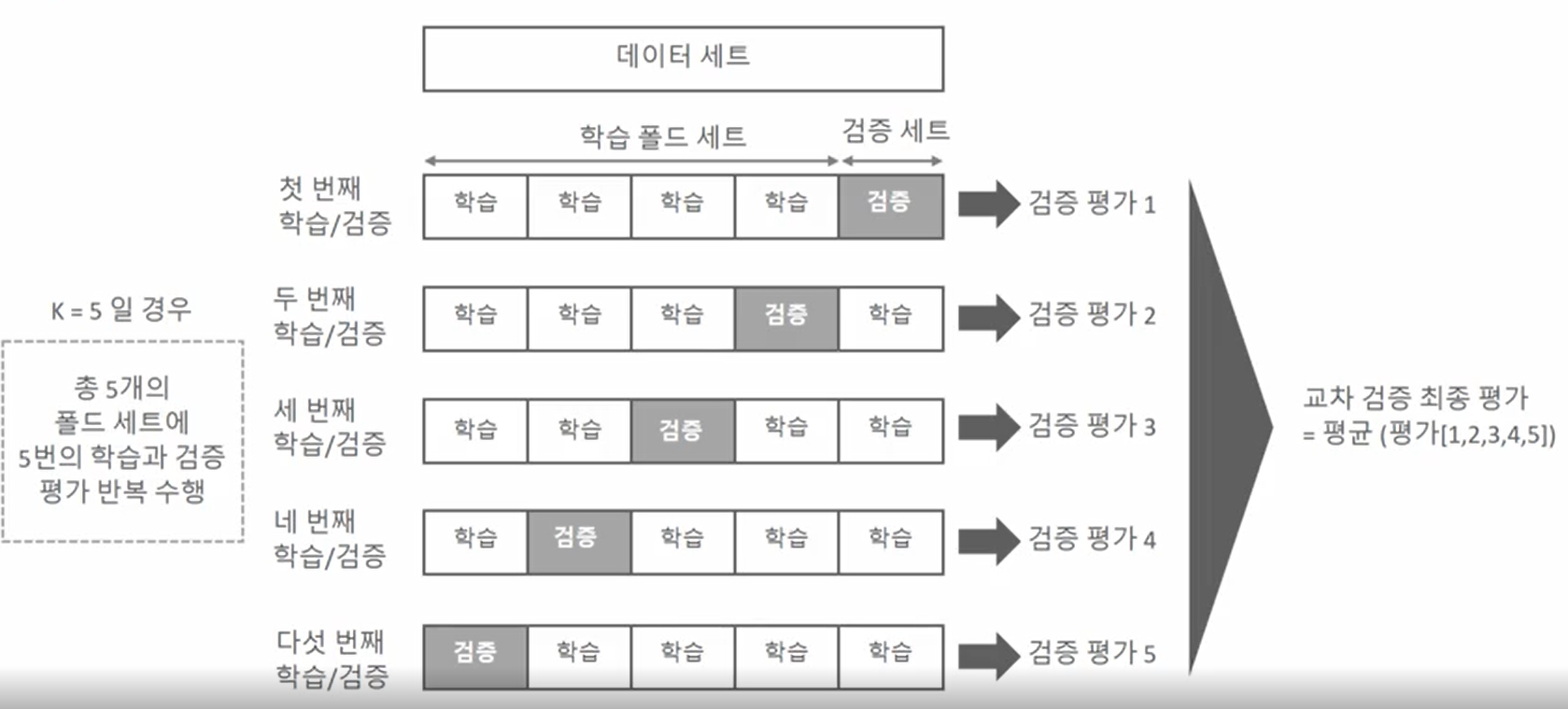
1) K 폴드 교차검증
K 폴드 교차검증은 가장 보편적으로 사용되는 교차검증 기법으로, K개의 데이터 폴드 세트를 만들어서 K번만큼 각 폴드 세트에 학습과 검증 평가를 반복적으로 수행하는 방법
→K개의 예측 평가를 구했으면 이를 평균해서 K 폴드 평가 결과로 반영
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import accuracy_score
from sklearn.model_selection import KFold
import numpy as np
iris = load_iris()
features = iris.data
label = iris.target
dt_clf = DecisionTreeClassifier(random_state=156)
# 5개의 폴드 세트로 분리하는 KFold 객체와 폴드 세트별 정확도를 담을 리스트 객체 생성
kfold = KFold(n_splits=5)
cv_accuracy = []
n_iter = 0 # for loop시 몇 번 반복했는지 확인하기 위해 n_iter 객체 생성
# KFold 객체의 split()를 호출하면 학습용, 검증용 테스트 데이터로 분할할 수 있는 인덱스를 array로 반환
for train_index, test_index in kfold.split(features):
# kfold.split()으로 반환된 인덱스를 이용해 학습용, 검증용 테스트 데이터 추출
x_train, x_test = features[train_index], features[test_index]
y_train, y_test = label[train_index], label[test_index]
# 학습 및 예측
dt_clf.fit(x_train, y_train)
pred = dt_clf.predict(x_test)
# 평가(정확도)
accuracy = np.round(accuracy_score(y_test, pred), 4)
# 폴드 세트별 정확도를 담을 리스트 객체에 정확도 추가
cv_accuracy.append(accuracy)
n_iter += 1
train_size = x_train.shape[0]
test_size = x_test.shape[0]
print('\n#{0} 교차검증 정확도:{1}, 학습 데이터 크기:{2}, 검증 데이터 크기:{3}'.format(n_iter, accuracy, train_size, test_size))
# split()이 어떤 값을 실제로 반환하는지 확인해 보기 위해 검증 데이터 세트의 인덱스도 추출해봄
print('#{0} 검증 세트 인덱스:{1}'.format(n_iter, test_index))
# 개별 iteration별 정확도를 합하여 평균 정확도 계산
print('\n## 평균 검증 정확도:', np.mean(cv_accuracy))
2) Stratified K 폴드
Stratified K 폴드는 불균형한 분포도를 가진 레이블 데이터 집합을 위한 K 폴드 방식
→Stratified K 폴드는 원본 데이터의 레이블 분포를 먼저 고려한 뒤 이 분포와 동일하게 학습과 검증용 테스트 데이터를 분배
StratifiedKFold를 사용하는 방법은 KFold를 사용하는 방법과 비슷하다. 단 하나 큰 차이는 StratifiedKFold는 레이블 데이터 분포도에 따라 학습/검증용 테스트 데이터를 나누기 때문에 split() 메서드에 인자로 피처 데이터 세트뿐만 아니라 레이블 데이터 세트도 반드시 필요함
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import accuracy_score
from sklearn.model_selection import StratifiedKFold
import numpy as np
iris = load_iris()
features = iris.data
label = iris.target
dt_clf = DecisionTreeClassifier(random_state=156)
# 3개의 폴드 세트로 분리하는 StratifiedKFold 객체와 폴드 세트별 정확도를 담을 리스트 객체 생성
skfold = StratifiedKFold(n_splits=3)
cv_accuracy = []
n_iter = 0 # for loop시 몇 번 반복했는지 확인하기 위해 n_iter 객체 생성
# StratifiedKFold 객체의 split()를 호출하면 학습용, 검증용 테스트 데이터로 분할할 수 있는 인덱스를 array로 반환
# StratifiedKFold의 split() 호출시 반드시 레이블 데이터 세트도 추가 입력 필요
for train_index, test_index in skfold.split(features, label):
# skfold.split()으로 반환된 인덱스를 이용해 학습용, 검증용 테스트 데이터 추출
x_train, x_test = features[train_index], features[test_index]
y_train, y_test = label[train_index], label[test_index]
# 학습 및 예측
dt_clf.fit(x_train, y_train)
pred = dt_clf.predict(x_test)
# 평가(정확도)
accuracy = np.round(accuracy_score(y_test, pred), 4)
# 폴드 세트별 정확도를 담을 리스트 객체에 정확도 추가
cv_accuracy.append(accuracy)
n_iter += 1
train_size = x_train.shape[0]
test_size = x_test.shape[0]
print('\n#{0} 교차검증 정확도:{1}, 학습 데이터 크기:{2}, 검증 데이터 크기:{3}'.format(n_iter, accuracy, train_size, test_size))
# split()이 어떤 값을 실제로 반환하는지 확인해 보기 위해 검증 데이터 세트의 인덱스도 추출해봄
print('#{0} 검증 세트 인덱스:{1}'.format(n_iter, test_index))
# 개별 iteration별 정확도를 합하여 평균 정확도 계산
print('\n## 평균 검증 정확도:', np.round(np.mean(cv_accuracy), 4))
그럼 KFold와 StratifiedKFold는 언제 써야 할까?
- KFold
- 회귀 문제(회귀의 결정값은 이산값 형태의 레이블이 아니라 연속된 숫자값이기 때문에 결정값 별로 분포를 정하는 의미가 없기 때문에 Stratified K 폴드가 지원되지 않음)에서의 교차 검증
- StratifiedKFold
- 레이블 데이터가 왜곡됐을 경우 반드시
- 분류 문제에서의 교차 검증
사이킷런은 교차검증을 좀 더 편리하게 수행할 수 있게 해주는 API를 제공함 → cross_val_score()
3) cross_val_score()

cross_val_score(estimator, X, y=None, scoring=None, cv=None, n_jobs=1, verbose=0, fit_params=None, pre_dispatch='2*n_jobs') → 이 중 estimator, X, y, scoring, cv가 주요 파라미터임
●estimator: 사이킷런의 분류 알고리즘 클래스인 Classifier 또는 회귀 알고리즘 클래스인 Regressor를 의미
●X: 피처 데이터 세트
●y: 레이블 데이터 세트
●scoring: 예측 성능 평가 지표
●cv: 교차검증 폴드 수
⇛cross_val_score()는 분류 문제인 경우 Stratified K 폴드 방식으로 작동하고, 회귀 문제인 경우 K 폴드 방식으로 작동
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import cross_val_score
import numpy as np
iris = load_iris()
features = iris.data
label = iris.target
dt_clf = DecisionTreeClassifier(random_state=156)
# 성능 지표는 정확도(accuracy), 교차검증 세트는 3개
scores = cross_val_score(dt_clf, features, label, scoring='accuracy', cv=3)
print('교차검증별 정확도:', np.round(scores, 4))
print('평균 검증 정확도:', np.round(np.mean(scores), 4))
4) 교차검증과 최적의 하이퍼파라미터 튜닝을 한번에 - GridSearchCV
사이킷런은 GridSearchCV를 이용해 분류와 회귀 알고리즘에 사용되는 하이퍼 파라미터를 순차적으로 입력하면서 편리하게 최적의 하이퍼파라미터를 도출할 수 있는 방안을 제공(최적의 하이퍼파라미터를 교차검증을 통해 추출)

<GridSearchCV의 주요 파라미터>
●estimator: 사이킷런의 분류 알고리즘 클래스인 Classifier 또는 회귀 알고리즘 클래스인 Regressor를 의미
●param_grid: key + 리스트 값을 가지는 딕셔너리가 주어짐. estimator의 튜닝을 위해 하이퍼파라미터명과 사용될 여러 하이퍼파라미터 값을 지정
●scoring: 예측 성능 평가 지표
●cv: 교차검증 폴드 수
●refit: 디폴트가 True이며 True로 생성시 최적의 하이퍼파라미터를 찾은 뒤, 입력된 estimator 객체를 해당 하이퍼파라미터로 재학습시킴
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import train_test_split, GridSearchCV
# 데이터를 로딩하고 학습 데이터와 테스트 데이터 분리
iris = load_iris()
features = iris.data
label = iris.target
x_train, x_test, y_train, y_test = train_test_split(features, label, test_size=0.2, random_state=121)
dtree = DecisionTreeClassifier()
# 파라미터를 딕셔너리 형태로 설정
parameters = {'max_depth':[1,2,3], 'min_samples_split':[2,3]}
grid_dtree = GridSearchCV(dtree, param_grid=parameters, scoring='accuracy', cv=3) # 디폴트로 refit=True
grid_dtree.fit(x_train, y_train)
# cv=3을 돌면서, 하이퍼파라미터 조합을 다 적용시켜서 그중 최적의 하이퍼파라미터 값
print('GridSearchCV 최적 파라미터:', grid_dtree.best_params_)
# cv=3을 돌면서, 하이퍼파라미터 조합을 다 적용시켜서 그중 최적의 하이퍼파라미터 값의 평가 결과 값
print('GridSearchCV 최고 정확도:', grid_dtree.best_score_)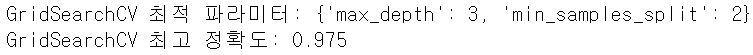
from sklearn.metrics import accuracy_score
# GridSearchCV의 refit=True로 최적의 하이퍼파라미터로 학습된 estimator 반환
estimator = grid_dtree.best_estimator_
# 최적의 하이퍼파라미터로 학습된 estimator를 이용해 테스트 데이터 세트에 대해 예측 및 평가
pred = estimator.predict(x_test)
print('테스트 데이터 세트 정확도:{0:.4f}'.format(accuracy_score(y_test, pred)))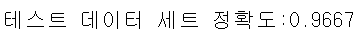
검증 세트의 최고 정확도 0.975보다 테스트 데이터 세트 정확도가 0.9667로 약간 낮게 나왔다. (검증 세트보다 약간 낮게 나오는게 일반적)→ 정확도 차이가 0.975보다 엄청 작게 나온게 아니므로 과적합이 없다고 판단
일반적인 머신러닝 모델 적용 방법은 학습 데이터를 GridSearchCV를 이용해 최적 하이퍼파라미터 튜닝을 수행한 뒤에 별도의 테스트 세트에서 이를 평가한다.
'파이썬 머신러닝 완벽가이드 > [2장] 사이킷런으로 시작하는 머신러닝' 카테고리의 다른 글
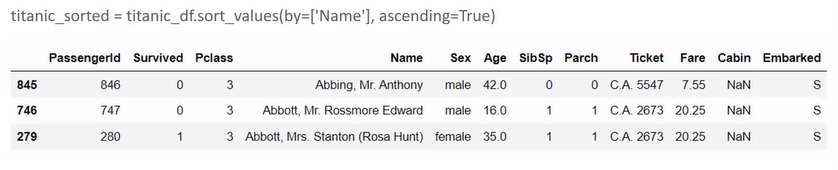
| 타이타닉 생존자 예측 실습 (0) | 2023.01.28 |
|---|---|
| 인코딩, 스케일링, 로그변환 익히기 (0) | 2023.01.25 |